聚类分析的核心依赖于样本间的距离或相似度度量,因此数据的尺度一致性、分布特性以及噪声处理直接决定了聚类结果的稳定性与可解释性。相比于监督学习,聚类对于数据质量更为敏感。本节将系统介绍在 MATLAB 环境下进行聚类前应完成的几项关键预处理任务。
2.1 数据读取与初步检查
% 读取模拟数据(你可将CSV导出自前一节数据)
T = readtable('simulated_ocean_data.csv');
% 初步查看数据结构
summary(T)确认数据完整性,检查是否存在缺失值、异常数值或维度错误。
Variables: SST: 300×1 double Values: Min 3.9547 Median 12.06 Max 19.852 Salinity: 300×1 double Values: Min 30.759 Median 33.47 Max 35.316 Chl_a: 300×1 double Values: Min 0.051747 Median 1.3826 Max 3.5635 TrueLabel: 300×1 cell array of character vectors
2.2 处理缺失值与不完整样本
在真实观测中,遥感或实测数据中常见部分缺失,处理策略包括删除或插值。
% 删除含缺失值的行(较安全)
T_clean = rmmissing(T);
% 如果仅少量缺失,也可按列均值填补
% for i = 1:width(T)-1
% T{isnan(T{:,i}), i} = mean(T{:,i}, 'omitnan');
% end2.3 特征标准化(Z-score 归一)
变量间存在单位与量纲差异(如 °C、PSU、mg/m³),聚类前必须统一尺度。推荐使用 Z‑score 标准化,即对每个变量减去均值、除以标准差。
% 取出特征数据(不含TrueLabel列)
X = T_clean{:, {'SST', 'Salinity', 'Chl_a'}};
% 执行Z-score标准化
X_norm = (X - mean(X)) ./ std(X);
% 可视化标准化前后的对比(可选)
figure;
subplot(1,2,1)
boxplot(X, 'Labels', {'SST', 'Salinity', 'Chl-a'});
title('原始特征分布');
subplot(1,2,2)
boxplot(X_norm, 'Labels', {'SST', 'Salinity', 'Chl-a'});
title('标准化后特征分布');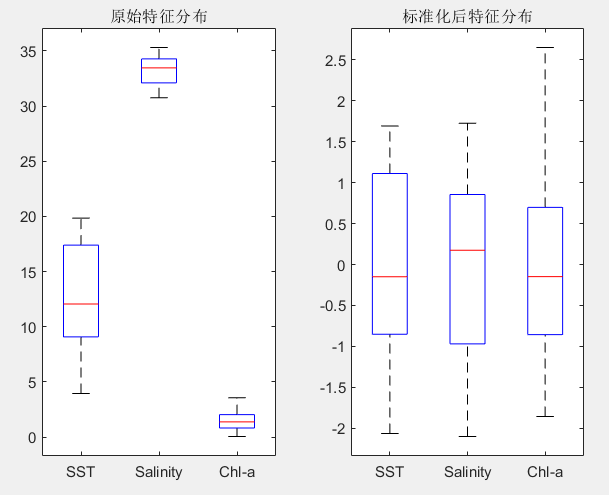
说明:Z-score 处理是基于欧氏距离的聚类方法(如 K-means、层次聚类)的前提条件。若后续使用基于密度或图结构的方法,仍建议统一尺度以增强数值稳定性。
2.4 异常值识别与剔除
极端值在聚类中可能被误识为独立簇,建议结合箱线图和 IQR 方法处理。
% 使用IQR剔除异常样本
Q1 = quantile(X_norm, 0.25);
Q3 = quantile(X_norm, 0.75);
IQR = Q3 - Q1;
% 定义上下限
lowerBound = Q1 - 1.5 * IQR;
upperBound = Q3 + 1.5 * IQR;
% 构建保留索引
idx = all(X_norm >= lowerBound & X_norm <= upperBound, 2);
X_filtered = X_norm(idx, :);
T_filtered = T_clean(idx, :);2.5 数据降维(可选)
如果特征维度超过 3–5,建议先进行主成分分析(PCA)降维,再聚类以消除冗余变量影响。此处示意三维数据下的降维可视化:
% 主成分分析(如需用于可视化)
[coeff, score, ~, ~, explained] = pca(X_filtered);
figure;
scatter(score(:,1), score(:,2), 40, 'filled')
xlabel(['PC1 (' num2str(explained(1), '%.1f') '%)'])
ylabel(['PC2 (' num2str(explained(2), '%.1f') '%)'])
title('PCA结果可视化(用于聚类前检查)')
grid on;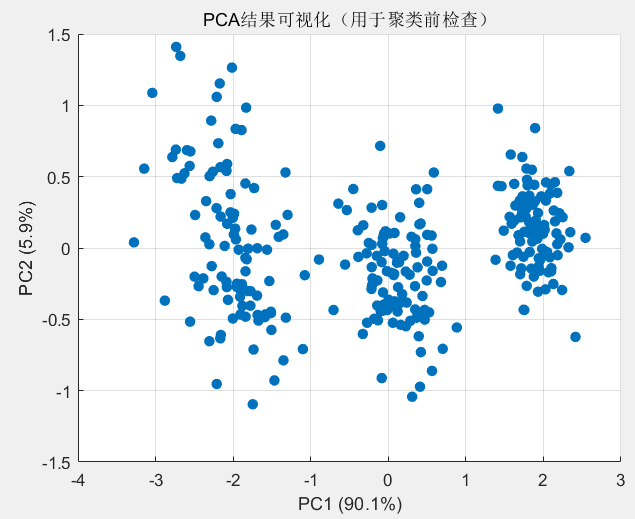
总结
本节以三变量模拟生态数据为例,在 MATLAB 中完成了以下数据准备步骤:
- 读取与检查数据结构
- 删除缺失值或插值填补
- 执行 Z‑score 标准化统一尺度
- 使用 IQR 检测并剔除异常值
- 降维以辅助可视化和稳健建模
